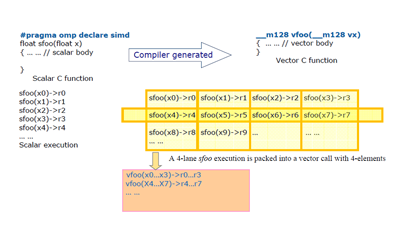
Accompagné techniquement par le Centre de compétences HPC@LR, le projet RCPG + GDP Release modélise les interactions entre les récepteurs couplés aux protéines-G et leurs partenaires afin d’aider au développement de nouveaux médicaments capables de les stimuler ou de les inhiber.
“Chez l’homme, les récepteurs couplés aux protéines-G (RCPG) forment une large famille de protéines trans-membranaires (plus de 800 membres) permettant à la cellule de communiquer avec son environnement. Ces récepteurs jouent un rôle important dans un grand nombre de processus physiologiques comme la perception visuelle, la douleur, la transmission nerveuse mais aussi dans la régulation de la pression artérielle ou de la prise alimentaire, etc.
De ce fait, le dérèglement d’un ou plusieurs de ces récepteurs a déjà été associé à de nombreuses maladies telles que le diabète, le cancer ou la schizophrénie. Pas moins de 40% des médicaments actuellement mis sur le marché ciblent un de ces récepteurs. On sait aujourd’hui que les RCPG ne fonctionnent jamais seuls : comme leur nom l’indique, ils sont couplés sur la face intra-cellulaire de la membrane plasmique à de grosses protéines partenaires, également associées à la membrane : les protéines-G. (cf Fig.1).
Des résultats récents montrent que sous l’effet d’un ligand extra-cellulaire, le plus souvent une petite molécule, ces récepteurs sont capables de changer de forme (ou conformation), et de transmettre cette information mécanique à la protéine-G, de l’autre côté de la bicouche de phospholipides. Dans son état inactif, la protéine-G est associée elle-même à une petite molécule, le GDP (guanosine di-phosphate). Sous l’effet de la stimulation mécanique produite par le récepteur, un échange GDP:GTP (guanosine tri-phosphate) va s’opérer au sein d’un des domaines de la protéine-G.“
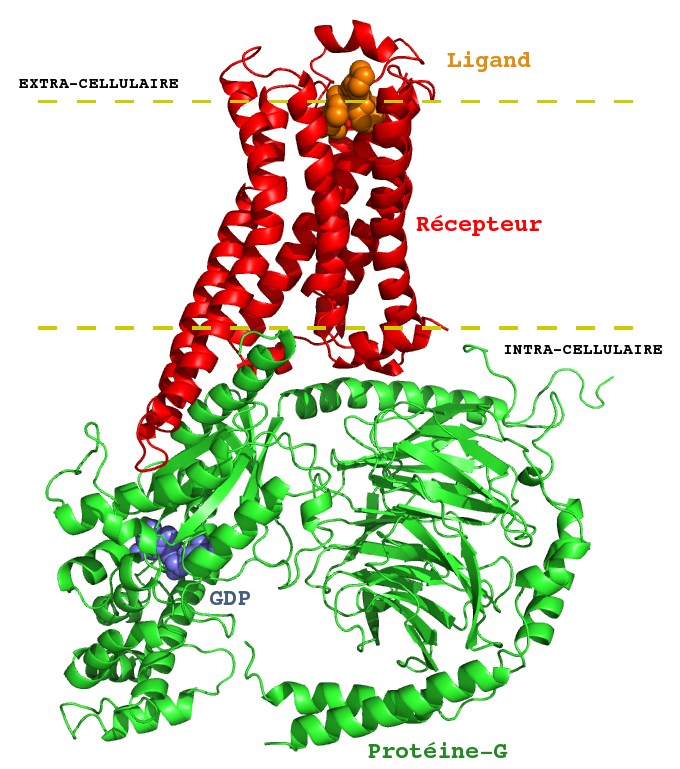
Interactions en chaîne
“De façon assez incroyable, la seule différence d’un phosphate entre le GDP et le GTP va conduire à des ré-arrangements structuraux, d’abord locaux, puis globaux, permettant la dissociation de la protéine-G en deux blocs. Chacun de ces blocs va pouvoir interagir à son tour avec d’autres protéines, assurant ainsi la propagation du signal à l’intérieur de la cellule (comme le montre en résumé cette vidéo). De cette façon, un message codé par la structure à l’échelle atomique d’une petite molécule extra-cellulaire est transmis mécaniquement à l’intérieur de la cellule, ce qui aboutit de proche en proche à une régulation de son fonctionnement.
Depuis quelques années, au sein du groupe de modélisation moléculaire de l’Institut des Biomolécules Max Mousseron (IBMM UMR5247), nous nous intéressons à la mécanique de ces récepteurs et de leurs protéines partenaires. Grâce au concours du Centre de compétences HPC@LR, nous avons d’ores et déjà proposé un mécanisme commun d’activation de ces récepteurs et des protéines-G associées.“
Des résultats très encourageants
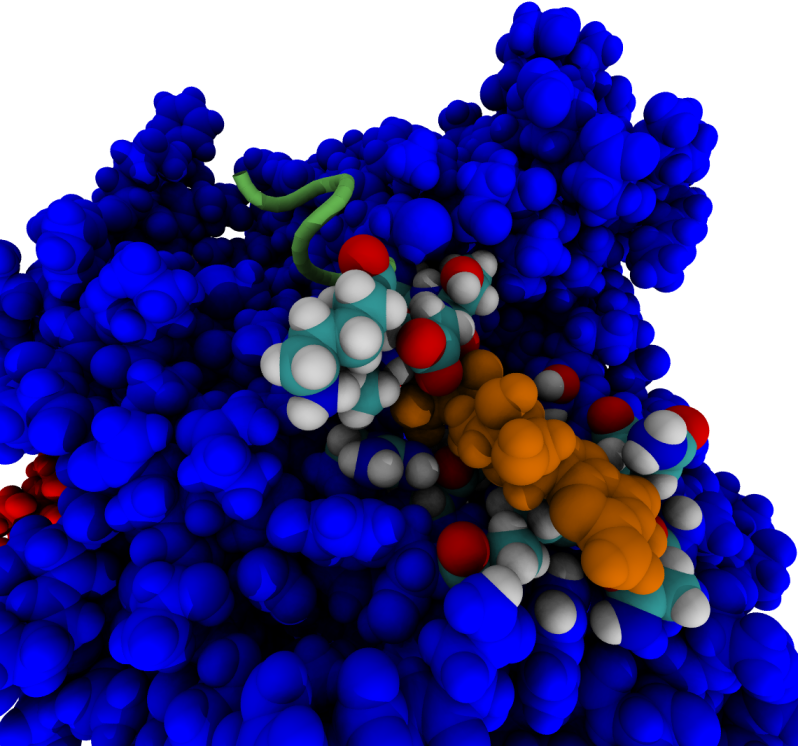
“En utilisant la dynamique moléculaire combinée à une méthode originale d’exploration des larges changements conformationnels des protéines, nous avons émis l’hypothèse que la sortie du GDP hors des protéines-G nécessitait un mouvement inter-domaine assez important (cf. Fig. 2). Ensuite, l’utilisation de méthodes plus quantitatives nous a permis de proposer une voie de sortie préférentielle pour le GDP et d’expliquer le rôle joué par certains résidus. Dernièrement, nous avons également montré comment la sortie du GDP et son remplacement par une molécule de GTP pouvait effectivement conduire à la dissociation de la protéine-G à la surface interne de la membrane plasmique.
Grâce à l’avènement récent et à la démocratisation des méthodes Gros-Grains (Coarse-Grained Molecular Dynamics), nous avons également pu proposer une orientation préférentielle de ces protéines à l’intérieur / à la surface des membranes biologiques. Des calculs sur les complexes protéine:protéine sont en cours dans ce domaine.
Avec ces modèles, et en collaboration avec nos collègues biochimistes et chimistes, nous espérons mieux comprendre la mécanique de ces récepteurs et de leurs partenaires afin d’aider au développement de nouveaux médicaments capables de les stimuler ou de les inhiber.“
Nicolas Floquet, Institut des Biomolécules Max Mousseron, Université Montpellier 1.
More around this topic...
© HPC Today 2024 - All rights reserved.
Thank you for reading HPC Today.